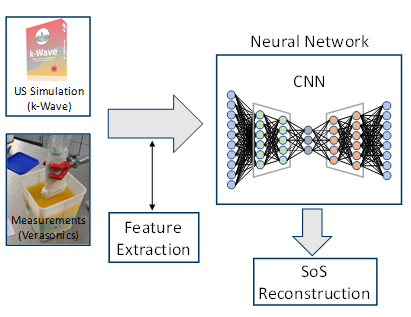
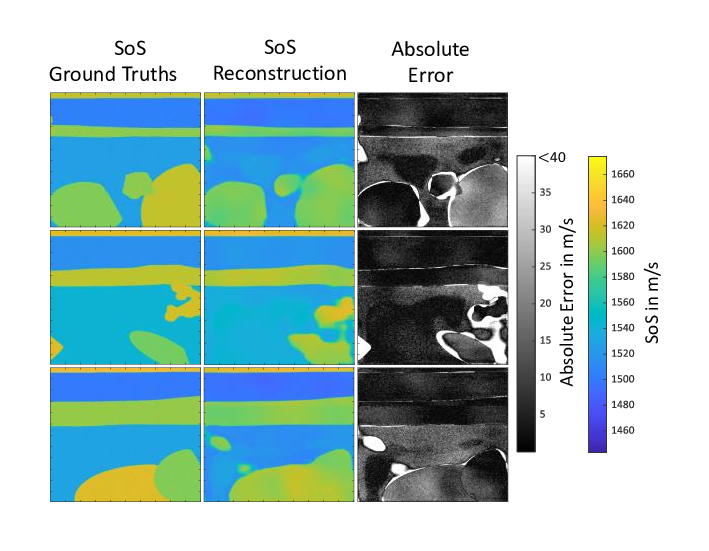
Die quantitative Rekonstruktion von Materialparametern mittels tomographischen Rekonstruktionverfahren wie der sogenannten Contrast Source Inversion oder durch das Kaczmarz Verfahren sind sehr rechenintensiv und zeitaufwändig. Der Zeitaufwand ist eine Folge daraus, dass ein inverses Problem gelöst werden muss, welches einerseits ein sogenanntes schlecht gestelltes sowie nichtlineares Problem darstellt. Zur Lösung wird bei diesen Verfahren auf ein zeitaufwändiges, iteratives Datenanpassungsverfahren zurückgegriffen. Als neuartiges, alternatives Rekonstruktionsverfahren, wird zur Zeit an neuartigen Deep-Learning basierten Methoden gearbeitet. Ein sogenannten Convolutional Neural Network wird hierzu an simulierten Ultraschalldaten, bzw. an einem aus diesen Daten extrahierten Kohärenzmaß, zusammen mit den zu rekonstruierenden Grundwahrheiten in Form der Schallgeschwindigkeitsverteilung trainiert.
Das Ziel besteht darin, dass das neuronale Netz den nichtlinearen Zusammenhang zwischen gemessenen Ultraschalldaten und der Materialparameterverteilung in Form der Schallgeschwindigkeit (SoS) erlernt. Anstelle der Verwendung von Ultraschallmessungen mit ringförmigen, das zu bildgebende Medium umschließenden Transducer-Aufbauten, die für klassische tomographische Rekonstruktionsverfahren nötig sind, sollen hierbei konventionelle lineare Ultraschallwandler verwendet werden. Herausforderungen bei diesem Ansatz liegen darin einen ausreichend großen und möglichst realistischen Trainingsdatensatz zu erzeugen und das Verfahren von der Anwendung auf Simulationsdaten auf die Anwendung an realen Messdaten zu transferieren.
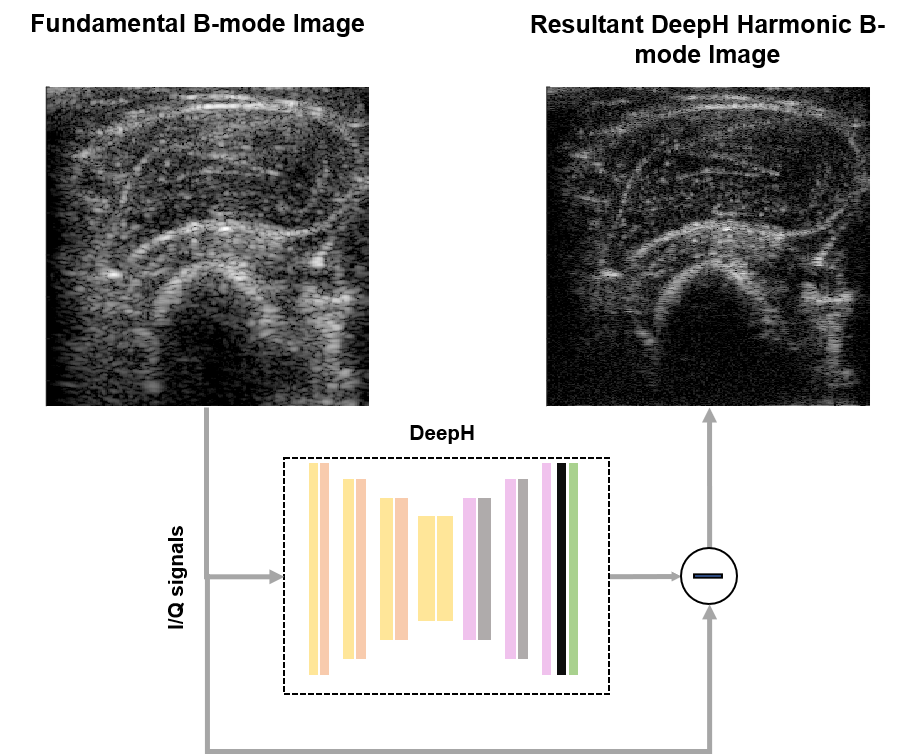
Tissue Harmonic Imaging (THI) ist ein bekanntes und wertvolles Bildgebungswerkzeug im klinischen Ultraschall, da es im Vergleich zur klassischen B-mode Bildgebung eine bessere Kontrastauflösung bietet und Bildartefakte erzeugt durch Mehrfachstreuungen unterdrückt. Die Trennung der harmonischen Signalanteile vom Gesamtsignal durch Nutzung von Hochpassfiltern kann allerdings aufgrund von Leck-Effekten zur Verschlechterung des Kontrastes oder zu einer geringeren axialen Auflösungen führen. Alternative nichtlineare Mehrimpulsverfahren für die harmonische Bildgebung wie Amplitudenmodulation und Impulsinversion leiden hingegen unter einer geringeren Bildrate und vergleichsweise höheren Bewegungsartefakten, da mindestens zwei Impulsechos erforderlich sind. Um dieses Problem zu lösen, wird am Lehrstuhl für Medizintechnik an einem Deep-Learning-basiertem Single-Shot-Harmonic-Imaging-Verfahren gearbeitet, das eine vergleichbare Bildqualität wie herkömmliche Methoden erzeugt, jedoch mit einer höheren Bildrate und weniger Bewegungsartefakten. Das Modell wurde für verschiedene Ziele und Proben evaluiert, um die Verallgemeinerbarkeit sowie die Möglichkeit und die Auswirkungen des Transferlernens zu veranschaulichen. Die neuartige Deep-Learning basierte Methode ist in der Lage mit einer einzigen Messung harmonische Bilder zu erzeugen. Die Bildqualität dieser Ultraschallbilder übertrifft die Qualität der durch lineare Filterung gewonnen Bilder und ist mit der Bildqualität von Multi-Puls-Erfassungen vergleichbar.
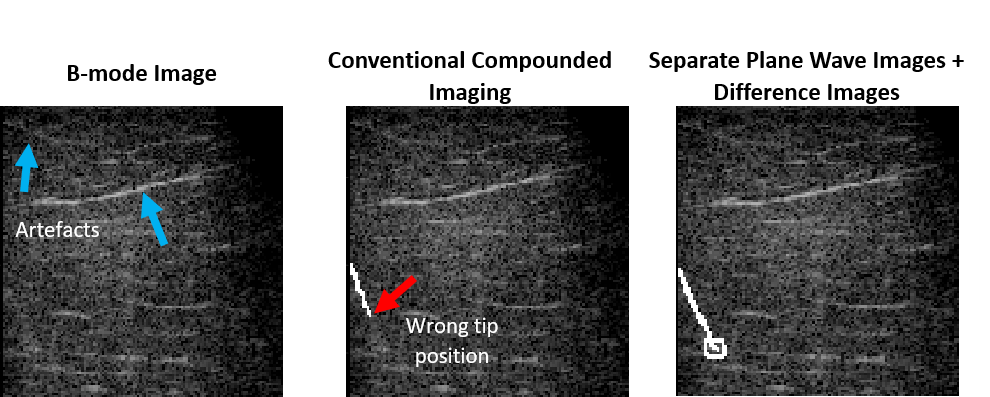
Die Sonographie wird üblicherweise zur Überwachung des Einstichs einer Kanüle in menschliches Gewebe verwendet. Insbesondere bei tiefen Einstichen und steilen Einstichwinkeln ist jedoch die Sichtbarkeit der Nadel in B-Mode Bildern selten zufriedenstellend. Am Lehrstuhl für Medizintechnik wird auf die Verwendung von DNNs zurückgegriffen um die nichtlinearen Informationen der injizierten Nadel zu verarbeiten. Anstelle die Information im finalen B-Mode Bild zu nutzen, die aus der Überlagerung mehrerer Messungen mit ebenen Wellen (engl. Plane Waves) unterschiedlichen Winkels entstehen, werden die Informationen in den Daten der Plane Wave Messungen direkt verwendet mit dem Ziel die Lokalisierung der Nadel zu verbessern. Aus diesem Grund wird ein Deep Learning basiertes Verfahren eingesetzt, das in der Lage ist, die Informationen aus den Einzelmessungen der verschiedenen Plane Wave Aufnamen zu extrahieren, um die Lokalisierung von Schaft und Spitze der Kanüle zu verbessern. Durch die Verwendung von separaten B-Mode Bildern, erstellt mit zusätzlichen Messungen mit ebenen Wellen, und deren Differenzbildern wird die Visualisierung der Nadel erheblich verbessert: Der Fehler bei der Lokalisierung der Spitze wird um das 4,5-fache verringert und die Schätzung des Einstichwinkels um 30,6 % verbessert, verglichen mit der direkten Verwendung von B-Mode Bildern, welche aus überlagerten Plane Wave Aufnahmen (engl. Compounded Imaging) erstellt wurden.